推荐产品
公司新闻/正文
Nature Biotechnology | 北京大学生命科学学院魏文胜课题组报道新型线粒体碱基编辑器
人阅读 发布时间:2023-05-24 16:01
线粒体作为细胞的「能量中心」在细胞生命活动过程中扮演着重要角色,它还是细胞核外存储遗传信息的另一细胞器。据 MITOMAP 数据库统计,在已确认的 97 种线粒体遗传疾病中,93 种由点突变引起,因此使用碱基编辑工具修正这些突变具有重要意义。然而线粒体中的电子传递将质子从线粒体基质排出,使得基质带有强负电荷,这阻碍了具有相同负电荷核酸(如 CRISPR 系统的 sgRNA)的进入。
全蛋白基础的基因编辑工具 ZFN 和 TALEN 可以在定位信号的引导下进入线粒体,之前有研究报道在小鼠线粒体中可以据此靶向敲低突变的基因组。然而,敲低线粒体基因组不能治疗纯合的线粒体突变,也不能主动改变线粒体基因组的碱基构成。
基于 CRISPR-Cas 系统开发的单碱基编辑在治疗基因组点突变引起的遗传疾病方面显示出巨大潜力。碱基编辑器中使用的脱氨酶都是单链 DNA 脱氨酶,在 Cas9 和 sgRNA 的作用下,目标 DNA 双链中的非靶向链暴露出来,为单链 DNA 脱氨酶实现有效碱基转换提供了必要条件。与 CRISPR-Cas 系统相比,锌指蛋白(ZF)和转录激活因子样效应子(TALE)只具有结合 DNA 双链的活性,却无法解开 DNA 双链。因此,简单将单链 DNA 脱氨酶与 ZF 或 TALE 结合无法实现对 DNA 的有效碱基编辑。
2020 年,Joseph Mougous 实验室和 David Liu 实验室利用一种能够作用于双链 DNA 的脱氨酶 DddA 开发出了线粒体单碱基编辑器,首次实现了线粒体基因 C->T 的碱基编辑。2022 年 Jin-Soo Kim 实验室使用 DddA 和 TadA8e 组合,进一步实现了线粒体基因组 A->G 的碱基编辑。然而,左二伟实验室和伊成器实验室随后发现 DddA 系统存在比较严重的脱靶效应,特别是 DddA 与 CTCF 存在相互作用,会产生细胞核基因组的非特异性编辑。
2023 年 5 月 22 日,北京大学魏文胜课题组在 Nature Biotechnology 杂志在线发表题为 「Strand-selective base editing of human mitochondrial DNA using mitoBEs」 的研究论文,报道了一种名为 mitoBEs 的全新线粒体单碱基编辑工具,该工具不依赖于 DddA 系统。mitoBEs 不仅能够高效地实现 A->G 或 C->T 的单碱基编辑,还具备选择性地编辑特定链的能力,这是 DddA 系统所不具备的。此外,研究人员未观察到明显的脱靶现象。
由于目前发现的除 DddA 之外的 DNA 脱氨酶,都只能作用于单链 DNA,整个研究基于这样的假设:在靶向位点产生瞬时的单链 DNA,可以为所有「普通」脱氨酶提供有效反应底物。因此,在 TALE 系统提供靶向的基础上,研究者整合了切口酶(nickase)和脱氨酶(deaminase),成功建立了高效的 TALE 版本的单碱基编辑器,实现了线粒体基因组的碱基编辑(mitochondrial DNA base editors, mitoBEs)。使用含有线粒体定位信号的 TALE-MutH 和 TALE-TadA8e(V106W) 靶向线粒体基因组,可以实现有效的 A->G 编辑(图 1)。
对切口酶 MutH 的突变研究,可以大幅度突破其序列偏好,将可编辑范围提高 10 倍以上。此外,无识别序列偏好的切口酶 Nt.BspD6I(C)和 FokI-FokI(D450A),也能够有效应用于 mitoBEs 系统(图 2)。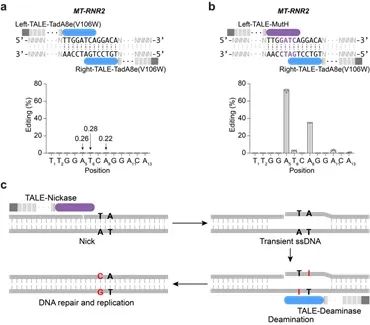
图 1: 引入 nickase 来实现链选择性 DNA 编辑及 mitoBE 工作模型
图 2: 筛选可以用于 mitoBE 系统的无识别序列限制的 nickase
除了 A->G 方向的编辑,TALE-MutH 和 TALE-rAPOBEC1-2×UGI 的结合可以实现 C->T 的高效碱基转换。与 DdCBE 相比,mitoBEs 具有链选择性偏好。此外,通过全基因组测序,mitoBEs 在线粒体和细胞核中都没有检测到严重的脱靶编辑,证明其具有高度特异性和安全性。
图 3: 使用 mitoBE 纠正 LHON 病人来源细胞的线粒体基因突变
研究者进一步尝试这一新型编辑器在疾病治疗中的应用。Leber 遗传性视神经病变 (Leber's hereditary optic neuropathy, LHON) 是一种由线粒体基因突变引起的急性眼部疾病,患者均为成年人。研究者利用环状 RNA 编码的 mitoBEs 实现了高效线粒体 DNA 链选择性碱基编辑,成功建立了疾病模型。
随后,针对 LHON 患者来源的细胞,使用环状 RNA 编码的 mitoBEs 在目标位点实现了约 20% 的编辑效率,修复后的细胞具有更高的 ATP 含量和氧化呼吸水平,表明 mitoBEs 对线粒体遗传疾病具有治疗潜力。这是首次通过碱基编辑方式修正了线粒体的致病突变(图 3)。由于理论上这一新技术能够修正大多数线粒体疾病突变(图 3),这就为治疗这些危害重大的疾病提供了有希望的治疗方法。此外,该技术方案也适用于细胞核基因组的碱基编辑,对相关疾病治疗提供了潜力巨大的新工具。
来源:北京大学生命科学学院
文章链接: https://doi.org/10.1038/s41587-023-01791-y
1.Gammage, P. A., Moraes, C. T. & Minczuk, M. Mitochondrial Genome Engineering: The Revolution May Not Be CRISPR-Ized. Trends Genet 34, 101-110, doi:10.1016/j.tig.2017.11.001 (2018).
2. Bacman, S. R. et al. MitoTALEN reduces mutant mtDNA load and restores tRNA(Ala) levels in a mouse model of heteroplasmic mtDNA mutation. Nat Med 24, 1696-1700, doi:10.1038/s41591-018-0166-8 (2018).
3 .Gammage, P. A. et al. Genome editing in mitochondria corrects a pathogenic mtDNA mutation in vivo. Nat Med 24, 1691-1695, doi:10.1038/s41591-018-0165-9 (2018).
4.Anzalone, A. V., Koblan, L. W. & Liu, D. R. Genome editing with CRISPR-Cas nucleases, base editors, transposases and prime editors. Nat Biotechnol 38, 824-844, doi:10.1038/s41587-020-0561-9 (2020).
5.Deng, D. et al. Structural basis for sequence-specific recognition of DNA by TAL effectors. Science 335, 720-723, doi:10.1126/science.1215670 (2012).
6.Yang, L. et al. Engineering and optimising deaminase fusions for genome editing. Nat Commun 7, 13330, doi:10.1038/ncomms13330 (2016).
7.Mok, B. Y. et al. A bacterial cytidine deaminase toxin enables CRISPR-free mitochondrial base editing. Nature 583, 631-637, doi:10.1038/s41586-020-2477-4 (2020).
8.Cho, S. I. et al. Targeted A-to-G base editing in human mitochondrial DNA with programmable deaminases. Cell 185, 1764-1776 e1712, doi:10.1016/j.cell.2022.03.039 (2022).
9.Wei, Y. et al. Mitochondrial base editor DdCBE causes substantial DNA off-target editing in nuclear genome of embryos. Cell Discov 8, 27, doi:10.1038/s41421-022-00391-5 (2022).
10.Lei, Z. et al. Mitochondrial base editor induces substantial nuclear off-target mutations. Nature 606, 804-811, doi:10.1038/s41586-022-04836-5 (2022).